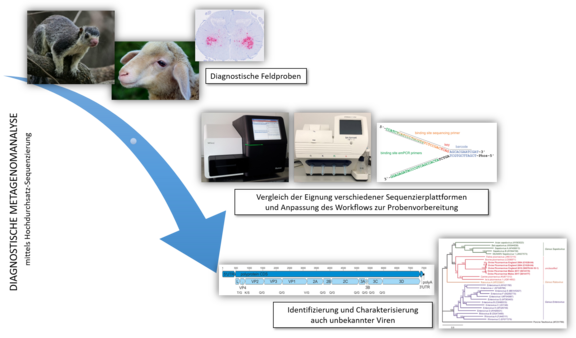
Die Möglichkeiten der Identifizierung von mikrobiellen Erregern bei unklarem Krankheitsgeschehen wurden durch den technischen Fortschritt im Bereich der Hochdurchsatz-Sequenzierung („next-generation sequencing“, NGS) enorm erweitert. Die darauf basierende diagnostische Metagenomanalyse ermöglicht heute, mit der Erfassung nahezu aller Nukleinsäuren einer klinischen Probe, eine ergebnisoffene Untersuchung und bietet damit zentrale Erkenntnisgewinne in Fällen, die mit etablierter Standard-Diagnostik nicht aufgeklärt werden können. Meine Promotionsarbeit trägt den Titel „Improved diagnostic metagenomics for virus discovery“ und beinhaltet zwei übergeordnete Schwerpunkte: Die Optimierung des Ablaufs der Probenvorbereitung für die Hochdurchsatz-Sequenzierung in Bezug auf diagnostische Anwendungen, sowie die Identifizierung und Charakterisierung unbekannter Viren bei unklaren Krankheitsgeschehen. Im Detail habe ich die Sequenziertechnologien von Illumina (Illumina MiSeq) und Life Technologies (Ion Torrent) anhand von Feldproben auf einen breiten Einsatz der Metagenomanalyse in der Diagnostik verglichen. Das vorteilhafte Konzept der Y-Adapter, das zur Probenvorbereitung bei der Illumina-Technologie Verwendung findet, habe ich auf Ion Torrent-Systeme übertragen und individuell zugeschnittene Y‑Adapter entworfen. Des Weiteren wurden die Eignung von Formalin-fixiertem und in Paraffin-eingebetteten Organen für die Diagnostik untersucht, wodurch das zur Untersuchung geeignete Probenspektrum erweitert werden konnte. Unter Anwendung dieser Optimierungen gelang es, zwei bis dahin unbekannte Viren zu entdecken und zu charakterisieren: Das Ovine Picornavirus, das bei jungen Lämmern eine tödliche Polioenzephalomyelitis hervorrufen kann und seit mindestens 20 Jahren in Großbritannien zirkuliert, und das Riesenhörnchen-Respirovirus, das bei einem in der Quarantäne-Station eines deutschen Zoos an einer akuten Pneumonie verstorbenen Riesenhörnchen identifiziert wurde. Die neu entdeckten Viren stellen aufgrund ihrer phylogenetischen Entfernung von bekannten Viren eigene Spezies dar und erweitern somit das Spektrum der bekannten viralen Erreger. Des Weiteren wurde die Methodik zur Aufklärung einer Erkrankungsreihe im Zusammenhang mit humanen Organtransplantationen angewendet, in der das bis zu dem Zeitpunkt umstrittene zoonotische Potenzial des klassischen Borna Disease Virus deutlich wurde.
Aus diesen Ergebnissen sind vier wissenschaftliche Veröffentlichungen entstanden (3x Erstautor, 1x geteilter Erstautor). Die Promotionszeit empfand ich als eine sehr intensive, lehrreiche und auch freudige Zeit, zu dem ein gutes Arbeitsumfeld und eine gute fachliche Betreuung zu beigetragen haben. Daher an dieser Stelle auch ein großes DANKE an alle, die mich durch diese Zeit begleitet haben und natürlich dem Förderverein des FLI für die Auszeichnung und Würdigung dieser Arbeit!
In der Doktorarbeit wurden diagnostische Feldproben, z.B. von einem Sri Lanka-Riesenhörnchen mit Pneumonie oder von jungen Lämmern mit Polioenzephalomyelitis, mittels einer Metagenomanalyse auf unbekannte Erreger untersucht. Zusätzlich wurden die Eignung der Sequenziertechnologien von Illumina (Illumina MiSeq) und Life Technologies (Ion Torrent) verglichen und zur Optimierung der Probenvorbereitung Y-Adapter entworfen. Abschließend wurden zwei bisher unbekannte Viren, das Ovine Picornavirus und das Riesenhörnchen-Respirovirus, entdeckt und charakterisiert.
Abb.: L. Forth
Foto Riesenhörnchen: verfügbar unter CC BY 2.0 von Steve Garvie (Dunfermline, Fife, Schottland)