Bekämpfung von Prionenkrankheiten in Tieren – Eine Genotypisierungs- und Prion-Stammcharakterisierungs-Studie
Prionenkrankheiten (Transmissible Spongiforme Enzephalopathien, TSEs) wie die Chronic Wasting Disease (CWD) in Zerviden, die klassische und atypische Bovine Spongiforme Enzephalopathie (BSE) im Rind sowie die klassische und atypische Scrapie der kleinen Wiederkäuer sind neurodegenerative Erkrankungen, die durch die Umfaltung des zellulären Prion-Proteins PrPC in die pathologische Isoform PrPSc verursacht werden. Während die BSE eine zoonotische Erkrankung ist, konnte dies für andere TSEs noch nicht eindeutig nachgewiesen werden. Darüber hinaus treten Prion-Proteine als sog. Prion-Stämme auf, die durch variable biologische Eigenschaften charakterisiert und vermutlich durch Konformationsunterschiede des Prion-Proteins bedingt sind. Ein wesentlicher Faktor, der Konformationsänderungen begünstigen kann, ist die PrPC-Struktur, die im Prion Protein Gen (PRNP) kodiert ist.
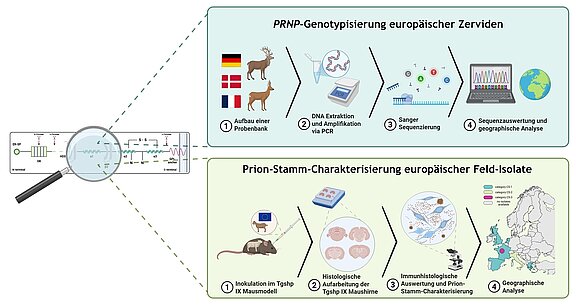
Daher wurde im ersten Teil dieser Dissertation, die PRNP-Variabilität in insgesamt 885 deutschen, 26 dänischen sowie 2114 französischen Proben von Reh-, Rot- und Sikawild untersucht. Während die Reh- und Sikawildproben homozygot waren, zeigte die Rotwildpopulation eine deutliche PRNP-Variabilität. So wurden insgesamt 14 Genotypen nachgewiesen, die zum Teil in geographischen Clustern auftraten. Nach Vergleichsanalysen mit nordamerikanischen und skandinavischen CWD-Studien, muss jedoch aktuell davon ausgegangen werden, dass die europäischen Zerviden vermutlich CWD-empfänglich sind. Prion-Stämme können durch Übertragungsexperimente in Tiermodellen charakterisiert werden. Um einen Überblick zu den in Europa zirkulierenden Prion-Stämmen zu erhalten, wurde im zweiten Teil der Dissertation eine Prion-Stammcharakterisierung im transgenen Mausmodell, dass das ovine Prion-Protein überexprimiert (Tgshp IX) durchgeführt. Das Probenpanel umfasste 31 klassische Scrapie Isolate sowie atypische Scrapie, caprine BSE (gtBSE) und den CH1641-Stamm als Referenzstämme, die mittels immunhistochemischer Untersuchung (PrPSc-Profil) der Tgshp IX-Mausgehirne eindeutig unterschieden werden konnten. Zudem gelang es, die klassischen Scrapie-Isolate in drei Prion-Stämme (CS-1 bis 3) zu kategorisieren.
Abbildung 1: Die Abbildung fasst die durchgeführten Untersuchungen bildlich zusammen. Im ersten Teil der Dissertation wurden Proben von europäischen Zervidenspezies untersucht um den Genotyp des Prion-Protein-Gens (PRNP) zu bestimmen. Nach Auswertung aller Daten erfolgte zusätzlich eine phylogeographischen Analyse, die insbesondere im Rotwild geographisch assoziierte PRNP-Cluster ergab. Der zweite Teil der Dissertation befasste sich mit der Prion-Stamm Charakterisierung europäischer Feld-Isolate mittels des transgenen Tgshp IX Mausmodells, das ovines Prion Protein überexprimiert. So konnten mindestens drei verschiedene Scrapie-Prion-Stämme in europäischen Ziegenpopulationen beschrieben werden. (Created in BioRender. Ernst, S. (2025) https://BioRender.com/qc0rr4q)
Sowohl die Untersuchung der PRNP-Genvarianten der Wirtspopulationen, als auch die Charakterisierung von Feld-Prion-Stämmen sind notwendige Informationen bei (neu) auftretenden Prion-Erkrankungen. Kontroll- und Tilgungsmaßnahmen können damit effektiv auch an potenziell zoonotische Prion-Stämme angepasst werden. Die sich aktuell stetig weiter ausbreitende CWD verdeutlicht die Notwendigkeit dieses Wissens.
Abschließend möchte ich mich ganz herzlich bei meinen Betreuer/innen, Kolleg/innen, Kooperationspartnern und Freund/innen am FLI für die tatkräftige Unterstützung während meiner Promotionszeit bedanken. Mein besonderer Dank gilt zudem den Mitgliedern des Fördervereins des Friedrich-Loeffler-Institutes für die Auszeichnung meiner Arbeit.